- 연구실식물유전자조절연구실
- 전공분자유전학
- 사무실 R1107, (연구실) R1108
- 전화02-705-8794
- 이메일byeongha@sogang.ac.kr
- 홈페이지http://pgrlab.sogang.ac.kr
학력 및 경력
1993. 2. SEOUL NATIONAL UNIVERSITY, Seoul, Korea, B.S. in Biology
1997. 2. SEOUL NATIONAL UNIVERSITY, Seoul, Korea, M.S. in Biology
2004. 5. UNIVERSITY OF ARIZONA, Tucson, USA, Ph.D. in Plant Sciences
2004. 5. ~ 2004. 7. UNIVERSITY OF ARIZONA, Tucson, USA, Postdoctoral Fellow
2004. 8. ~ 2007.7. COLD SPRING HARBOR LABORATORY, Cold Spring Harbor, USA, Postdoctoral Fellow
2007. 9. ~ 현재 SOGANG UNIVERSITY, Seoul, Korea, Assistant Professor, Associate Professor, Professor
개설 강의
· 식물발달생물학 (Plant Developmental Biology)
· 식물생명과학 (Plant Biology)
· 분자생물학 (Molecular Biology)
연구 분야
(1) RNA 생물학, (2) 발달생물학, (3) 스트레스 생물학
<연구내용>
(1) mRNA processing 과 microRNA 생성
애기장대 STABILIZED1 (STA1) 유전자는 pre-mRNA splicing factor인 PRP6 유사단백질을 암호화하고 있고 mRNA splicing과 stability에 관여하고 있다. 우리는 STA1 유전자가 splicing 기능을 통해서 microRNA 생성에도 관여함을 밝혔다. STA1 유전자에 결함이 발생한, 애기장대 sta1-1 돌연변이체는 microRNA 축적이 정상체에 비해서 현저히 낮고, microRNA가 표적하는 전사체의 축적이 증가했다. 또한 sta1-1 돌연변이체는 splicing이 안 된 pri-miRNA 축적이 증가했다. 이에 우리는 이 연구를 확장하기 위해서 STA1을 중심으로 하는, mRNAprocessing/splicing과 microRNA 생성 상호작용 기작을 중점적으로 연구하고 있다. 이를 위해 sta1-1 표현형이 증가된 enhancer 돌연변이체와 표현형이 감소된 suppressor 돌연변이체를 확보하였고, 이들에서 STA1과 유전학적으로 상호작용하는 유전자를 찾고 있다.
STA1이 splicing factor이기 때문에, 세포내에서 항상적으로 필요할 것 같음에도, 이러한 온도 스트레스에 의해 그 발현이 유도된다. 이에, STA1 발현을 조절하는 유전자를 찾고 있다. Yeast one-hybrid 기법을 이용해서 STA1 프로모터에 결합하는 두 전사인자를 분리하였고 이에 대한 특징과 기능을 분석하고 있다. 또한 STA1 프로모터에 luciferase를 결합한 리포터 유전자를 가지는 애기장대를 이용하여 STA1 발현이 변화한 돌연변이체를 분리하였고 이 돌연변이체의 특징과 그 돌연변이를 일으키는 유전자, 즉 STA1 발현을 조절하는 유전자를 찾고 있다.
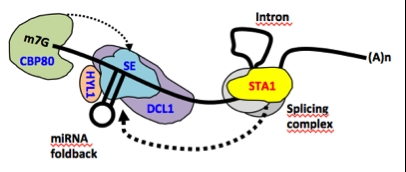
STA1의 microRNA 생성 관여 모델 Yeast one-hybrid를 통해 분리된 STA1프로모터 결합 전사인자
(2) 식물 발달프로그램
Altered Meristem Program1 (AMP1) 유전자는 glutamate carboxypeptidase를 암호화하고 있다. AMP1 유전자에 돌연변이가 발생한 애기장대 amp1-1 돌연변이체는 정상체에 비해서 잎을 더많이 생성하고 이에 따라 꽃을 빨리 피운다. 우리는 최근 AMP1이 종자 발아 형태에도 관여함을 밝히고 이들 표현형을 식물 호르몬 신호전달을 통해 설명하기 위한 연구를 하고 있다.
또 다른 식물 형태 결함 돌연변이체인 small and thick leaf 1 (stl1) 돌연변이체는 이름이 말해주듯이 작고 두꺼운 잎을 가지고 있다. 이 돌연변이체는 또한 잎 표면의 왁스층에 더 많은 지질(lipid)을 축적하고 있다. 우리는 최근 이 돌연변이 형태를 유발하는 돌연변이 유전자를 찾았고 이 유전자의 기능을 연구하고 있다. 특히, 이 돌연변이체에서 식물의 줄기세포의 하나라고 할 수 있는 형성층이 증가한 것을 관찰하여, 이 STL1 유전자의 식물 형성층 줄기세포에서의 기능 연구에 초점을 맞추고 있다.


정상체 Col-0와 amp1-1표현형비교 정상체 (C24RDLuc)과 stl1표현형 비교
(3) 식물 스트레스 신호전달과 대응 기작: 유전자가위의 활용
C-repeatbinding protein (CBF)는 식물 저온 스트레스 신호전달에서 중요한 전사인자다. 애기장대에는 저온 스트레스 신호전달에 관여하는 CBF 전사인자가 3개 존재하고 각각 CBF1, CBF2, CBF3라고 불린다. 우리는 CBF3 프로모터에 luciferase 유전자를 결합한 리포터 시스템을 이용해서 CBF3 발현이 변화한 돌연변이체를 찾았다. 이 중 하나인 highly induced CBF3 expression1 (hic1)는 저온에서 정상체보다 더 높은 CBF3 발현을 보인다. 우리는 이 hic1 돌연변이체의 저온 스트레스를 포함한 각종 스트레스 조건에서의 반응성을 분석하고 있으며, 이런 돌연변이 표현형을 일으키는 원인이 되는 결함 유전자를 찾고 있다. 이 유전자를 찾게 되면 결국 CBF3 유전자발현 조절에 중요한 유전자를 찾게 되는 셈이 된다.
또한 우리는 유전자가위인 CRISPR/Cas 시스템을 이용하여 애기장대의 CBF1, CBF2, CBF3 유전자를 모두 제거했다. 특히, 애기장대의 각 다른 아종이라고 할 수 있는 Columbia-0, C24, Landsberg erecta에서 모두 CBF1, 2, 3 유전자를 삭제하여 각 다른 애기장대에서 저온신호전달을 연구할 수 있는 토대를 만들었다.
아울러, 극지연구소와 공동연구로, 남극에 존재하는 이끼에서 분리한 유전자를 애기장대에 과발현시켜 더 강한 스트레스 저항성을 갖게 하는 유전자를 찾고 있다. 이미 이에 해당하는 극지 이끼 유전자를 수 종 확보하였다. 우리는 이와 같은 극지연구소와의 공동연구를 통해서 극지 유전자원의 활용도를 높일 수 있을 것으로 기대한다.
우리 연구실은 국내에서 CRISPR/Cas 유전자 가위를 식물에 적용한 선두 그룹으로 이를 스트레스 저항성 식물 연구 및 활용에 적극 적용하려고 한다.

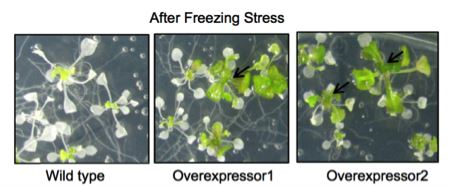
hic1의 높은 CBF3-LUC luminescence 발현 극지이끼유전자 과발현체의 저온스트레스 저항성 증가
연구 업적
1. Yolcu, S., Alavilli, H., Lee, B.-h. (2020) Natural Genetic Resources from Diverse Plants to Improve Abiotic Stress Tolerance in Plants. Int J Mol Sci. 21(22):8567. doi: 10.3390/ijms21228567.
2. Lim CJ, Park J, Shen M, Park HJ, Cheong MS, Park KS, Baek D, Bae MJ, Ali A, Jan M, Lee SY, Lee, B.-h., Kim WY, Pardo JM, Yun DJ. (2020) The Histone-Modifying Complex PWR/HOS15/HD2C Epigenetically Regulates Cold Tolerance. Plant Physiol. 184:1097-1111. doi: 10.1104/pp.20.00439.
3. Hwang, J.-H., Yu, S.-i., Lee, B.-h.* and Lee, D.-H.* (2020) Modulation of energy metabolism is important for low-oxygen stress adaptation in Brassicaceae species. Intl J. Mol. Sci. 21: 1787. doi: 10.3390/ijms21051787. * Co-corresponding author.
4. Yu, H., Kong, X., Huang, H., Wu, W., Park, J., Yun, D.-J., Lee, B.-h., Shi, H., Zhu, J.-K. (2020) STCH4/REIL2 confers cold stress tolerance in Arabidopsis by promoting rRNA processing and CBF protein translation. Cell Rep. 30: 229-242. doi: 10.1016/j.celrep.2019.12.012.
5. Kim, H., Yu, S.-i., Jung, S.H., Lee, B.-h.* and Suh, M.-C.* (2019) The F-box Protein SAGL1 and ECERIFERUM3 Regulate Cuticular Wax Biosynthesis in Response to Changes in Humidity in Arabidopsis. Plant Cell 31: 2223-2240. doi:10.1105/tpc.19.00152. * Co-corresponding author.
6. Tong, X., Oh, E.K., Lee, B.-h., and Lee J.K. (2019) Production of long-chain free fatty acids from metabolically engineered Rhodobacter sphaeroides heterologously producing periplasmic phospholipase A2 in dodecane-overlaid two-phase culture. Microbial Cell Factories 18:20. Doi: 10.1186/s12934-019-1070-8.
7. Yu, S.-i., Kim, H., Yun, D.-J., Suh, M.C., and Lee, B.-h.* (2019) Post-translational and transcriptional regulation of phenylpropanoid biosynthesis pathway by Kelch repeat F-box protein SAGL1. Plant Molecular Biology 99: 135-148. doi: 10.1007/s11103-018-0808-8.
8. Park, M., Hong, S.G., Park, H., Lee, B.-h.*, Lee, H.* (2018) Identification of reference genes for RT- qPCR in the Antarctic moss Sanionia uncinata under abiotic stress conditions. PLoS ONE 13(6): e0199356.
9. Park, J., Lim, C.J., Shen, M., Park, H.J., Cha, J.-Y., Iniesto, E., Rubio, V., Mengiste, T., Zhu, J.-K., Bressan, R.A., Lee, S.Y., Lee, B.-h., Jin, J.B., Pardo, J.M., Kim, W.Y., and Yun, D.J. (2018) Epigenetic switch from repressive to permissive chromatin in response to cold stress. Proc. Natl. Acad. Sci. U. S. A. doi: 10.1073/pnas.1721241115.
10. Alavilli, H., Lee, H., Park, M., Yun, D.-J., and Lee, B.-h. (2018). Enhanced multiple stress tolerance in Arabidopsis by overexpression of the polar moss peptidyl prolyl isomeraseFKBP12 gene. Plant Cell Reports 37: 453-465. doi: 10.1007/s00299-017-2242-9.
11. Park, M., Park. H., Lee, H.*, Lee, B.-h.*, and Lee, J.* (2018). The Complete Plastome Sequence of an Antarctic Bryophyte Sanionia uncinata (Hedw.) Loeske. International Journal of Molecular Sciences 19: 709. doi:10.3390/ijms19030709
12. Cho, S., Yu, S.-i., Park, J., Mao, Y., Zhu, J.-K., Yun, D.-J., and Lee, B.-h. (2017). Accession-Dependent CBF gene deletion by CRISPR/Cas system in Arabidopsis. Front. Plant Sci. 8: 1910. doi: 10.3389/fpls.2017.01910.
13. Yu, S.-i., and Lee, B.-h. (2017). Generation of a stress-inducible luminescent Arabidopsis and its use in genetic screening for stress-responsive gene deregulation mutants. In Plant Stress Tolerance: Methods and Protocols, R. Sunkar, ed (New York, NY: Humana Press/Springer Nature), pp. 109-119.
14. Kown, S.-J., Kown, O.-S., Kim, K.-T., Go, Y.-H., Yu, S.-i., Lee, B.-h., Miyoshi, H., Oh, E., Cho, S.-J., Cha, H.-J. (2017). Role of MEK partner-1 in cancer stemness through MEK/ERK pathway in cancerous neural stem cells, expressing EGFRviii. Mol. Cancer 16: 140.
15. Alavilli, H., Lee, H., Park, M., and Lee, B.-h. (2017). Antarctic moss Multiprotein Bridging Factor 1c overexpression in Arabidopsis resulted in enhanced tolerance to salt stress. Front. Plant Sci. 8, 1206. doi: 10.3389/fpls.2017.01206.
16. Hwang, S.-T., Li, H., Alavilli, H., Lee, B.-h., and Choi, D. (2017). Molecular and physiological characterization of AtHIGD1 in Arabidopsis. Biochem. Biophys. Res. Com. 487, 881-886.
17. Kim, G.-D., Cho, Y.-H., Lee, B.-h., and Yoo, S.-D. (2017). STABILIZED1 modulates pre-mRNA splicing for thermotolerance. Plant Physiol. 173: 2370.


